date: 2025-01-07
title: CF-DNA-Data
status: DONE
author:
- AllenYGY
tags:
- Data
publish: falseData
replication time domain,
甲基化; Genome-wide DNA methylation analysis
Open chromatin profiling with ATAC-seq
Chromatin modifications profiling with Chip-seq; peak
/active regulatory elements
eQTL?
Gene expression
DNA repair related???
Clearance Mechanisms?
repetitive regions (e.g., satellite DNA or LINE/SINE elements)
GC contentGO biology process???
Replication Time Domain(复制时间域)
甲基化与全基因组 DNA 甲基化分析(Genome-wide DNA Methylation Analysis)
- 甲基化:是指在 DNA 分子中添加甲基基团(通常在 CpG 位点)。这种表观遗传修饰能够调控基因表达。
- 全基因组甲基化分析:通过技术(如 Bisulfite-Seq)检测整个基因组范围内的 DNA 甲基化模式,用于研究基因调控、疾病机制等。
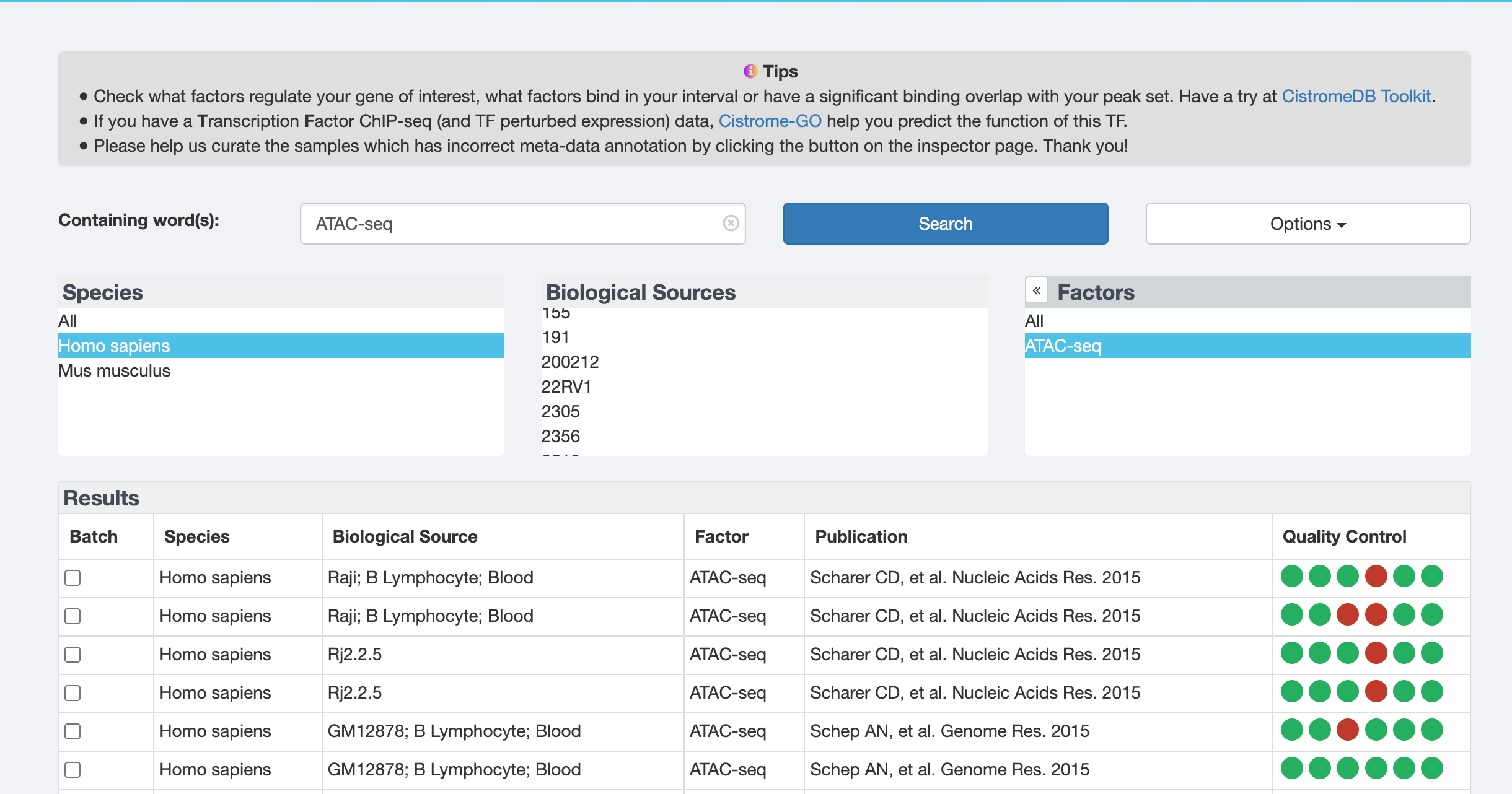
开放染色质分析(ATAC-seq)
- ATAC-seq(Assay for Transposase-Accessible Chromatin with high-throughput sequencing):是一种测定染色质可及性的方法,通过检测转座酶能插入的开放区域,确定潜在活跃的调控区域(如启动子、增强子)。
- 应用:识别细胞中基因活跃调控区域,研究基因调控网络。

GRCh38
ENCSR485TLP
ENCSR032RGS
...
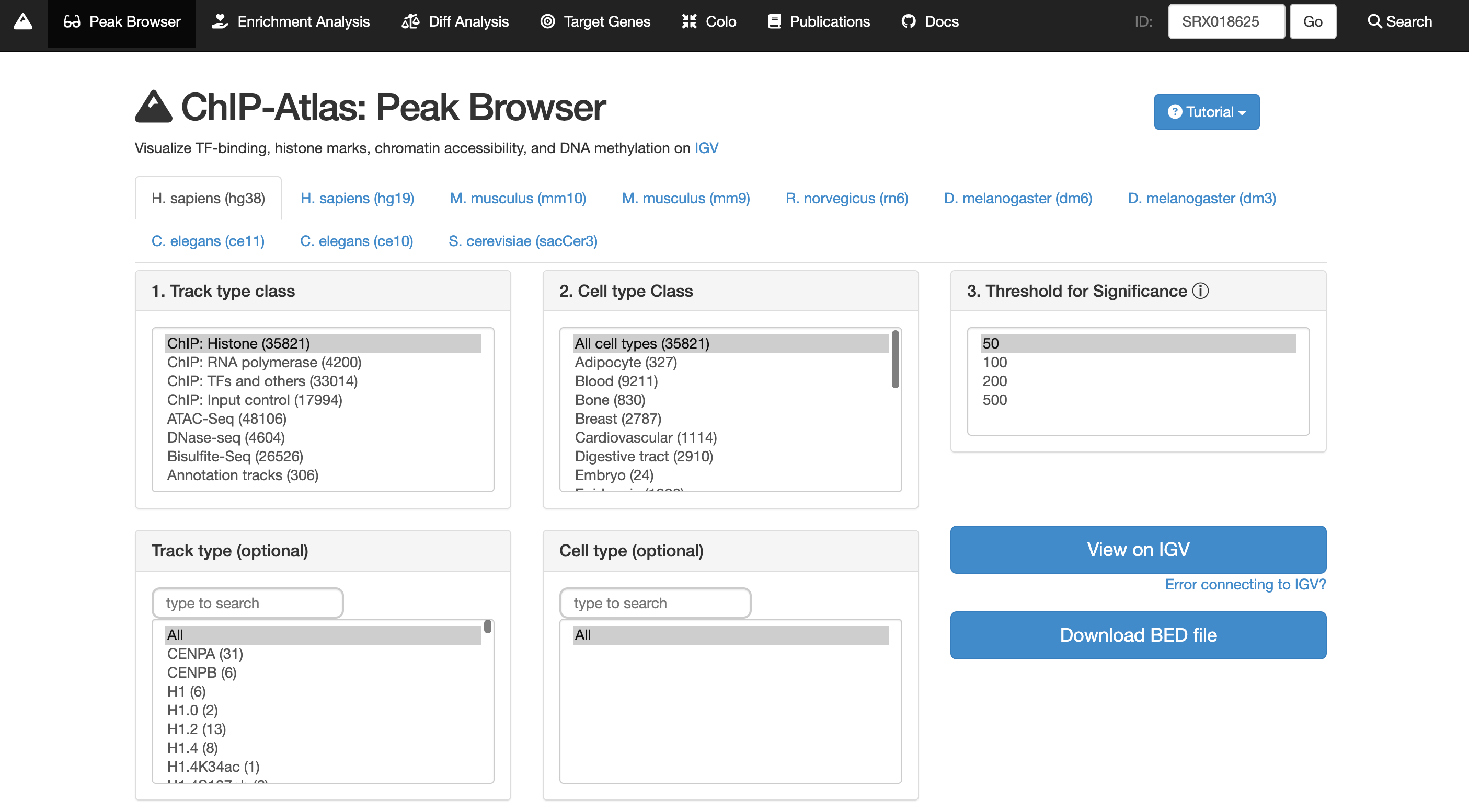
染色质修饰分析(ChIP-seq)与 Peaks 和活跃调控元件
- ChIP-seq(Chromatin Immunoprecipitation Sequencing):用于研究特定蛋白(如转录因子或修饰酶)与染色质的相互作用。
- Peaks:代表 ChIP-seq 数据中检测到的蛋白质与 DNA 结合的高峰区域,通常对应活跃的调控元件(如启动子或增强子)。
- 活跃调控元件:经常表现为开放染色质,结合活跃的转录因子,驱动基因表达。
eQTL(Expression Quantitative Trait Loci)
- eQTL 是一种基因组区域,控制基因表达水平的变异。
- 类型:
- Cis-eQTL:调控位点靠近目标基因。
- Trans-eQTL:调控位点远离目标基因。
- 应用:用于理解遗传变异如何影响基因表达,从而影响表型和疾病。
DNA Repair Related
- DNA 修复机制:细胞用来修复 DNA 损伤(如双链断裂、碱基错配等)的生物学过程。常见途径包括:
- 碱基切除修复(BER)
- 核苷酸切除修复(NER)
- 同源重组修复(HR)
- 非同源末端连接(NHEJ)
Clearance Mechanisms
- 指细胞清除不需要的分子或受损结构的过程。
- 示例:
- 自噬(Autophagy):清除受损细胞器或蛋白质。
- 蛋白酶体介导的降解。
- 核酸酶清除受损 DNA。
Repetitive Regions
- 卫星 DNA(Satellite DNA):存在于着丝粒或端粒的重复序列,通常参与染色体结构维护。
- LINE/SINE 元件:长散布元件(LINE)和短散布元件(SINE)是转座因子的一部分,可能影响基因组的稳定性。
GC 含量(GC Content)
- 基因组序列中鸟嘌呤(G)和胞嘧啶(C)碱基的比例。
- 意义:
- 高 GC 含量区域通常与基因密集区相关。
- 影响 DNA 复制、转录和结构稳定性。
Gene Ontology (GO) 生物过程(Biological Process)
- Gene Ontology (GO):一个标准化术语集,用于描述基因的功能。
- Biological Process:描述基因在生物学过程中的作用,例如信号传导、细胞分裂或代谢。
Table Of Contents